商机详情 -
青海合成噬菌体文库流程解析
Fab由重链Fd片段和完整轻链通过二硫键连接。构建Fab合成抗体库常利用人工设计的CDR序列融合于可变区框架形成Fab抗体基因并插入噬菌体的外壳蛋白基因,融合蛋白表达于噬菌体表面即可展示多样性的抗体。经过多轮噬菌体与抗原结合、洗脱和扩增后所得的抗体即为所需的特异性抗体。人工设计的过程中对所需抗体的基因序列进行修饰以去除影响特异性和稳定性的氨基酸片段,这样筛选出的抗体通常成药性更好,亲和力更高。Fab抗体基因上CDR序列完全由人为突变或随机化设计而表达的抗体称为全合成抗体库,该类抗体库通常容量更大,能筛选出针对特定靶点而天然免疫反应难以生产的抗体。当通过其他方式筛选获得抗体的性能不佳时,在天然抗体的基础上人工突变某些位点建库以筛选出符合性状的抗体,以此方式构建的抗体库属于半合成抗体库。半合成抗体库通常用于改善天然抗体的亲和力,生物/物理性状以及对抗体进行人源化改造。全人源Fab合成噬菌体文库,上海溪长技术加持,研究更轻松。青海合成噬菌体文库流程解析
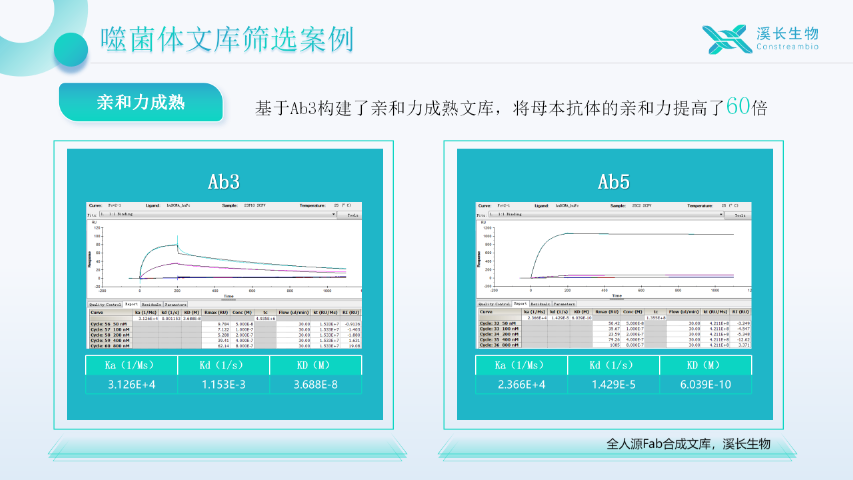
在抗体药物研发领域,亲和力是衡量抗体与抗原结合能力的关键指标,直接决定药物的疗效与成药潜力。上海溪长生物技术有限公司的全人源Fab合成噬菌体文库,凭借分子设计-文库构建-筛选优化全链条技术创新,将抗体亲和力提升至皮摩尔(pM,10⁻¹²M)级别,较传统naïve库提升100-1000倍,接近理论上抗原-抗体结合的“完美匹配”状态,为攻克自身免疫性疾病等重大疾病提供了前所未有的高效武器。经全人源Fab合成文库筛得的抗体亲和力水平高,经数十个靶点验证,确保药物候选分子一步到位。在筛选效率上,采用‘筛选-优化-交付’全流程自动化系统,2周内完成从靶点对接至高亲和力克隆交付,较传统方法缩短70%周期,抢占研发黄金窗口。中国台湾合成噬菌体文库服务抗体研究新方向,上海溪长全人源Fab合成噬菌体文库,资源丰富前景广。

上海溪长生物的全人源Fab合成文库是抗体研发领域的创新典范。在构建文库时,充分考虑了抗体研发的实际需求,采用了一系列先进技术。例如,在选择germline序列时,经过大量的实验和数据分析,挑选出了适合噬菌体展示、表达量高且成药性好的序列。随机化策略方面,选用trimer引物,精心设计CDR3区域,使其长度和氨基酸分布呈现多样化,极大地丰富了文库的复杂性。这种精心设计的文库,在筛选抗体时具有极高的效率和准确性。无论是针对新发现的疾病靶点,还是对现有抗体进行优化升级,全人源Fab合成文库都能发挥重要作用。目前,溪长生物已与众多科研机构和药企建立了合作关系,通过该文库为他们提供了高水平的抗体筛选服务,助力多个研发项目取得了阶段性成果,在行业内树立了良好的口碑。
随着生命科学研究的不断深入,越来越多的新兴靶点被发现,这些靶点往往与一些难治性疾病的发展密切相关。然而,传统的抗体筛选方法在面对这些新兴靶点时,常常面临挑战。全人源Fab合成文库凭借其丰富的抗体多样性和高效的筛选机制,能够迅速响应新兴靶点的筛选需求。即使对于那些全新的、研究较少的靶点,文库也能够通过筛选,找到与之特异性结合的抗体。这一优势为科研机构和药企在探索未知疾病领域、开发新型治疗方法方面提供了关键支持。目前,溪长生物已成功利用该文库筛选出针对多个新兴靶点的抗体,为相关疾病的研究和治疗开辟了新的途径。选择上海溪长生物的全人源Fab合成文库,为攻克更多疑难病症贡献力量。上海溪长全人源Fab合成噬菌体文库,多源整合,为抗体研究提供可靠支撑。
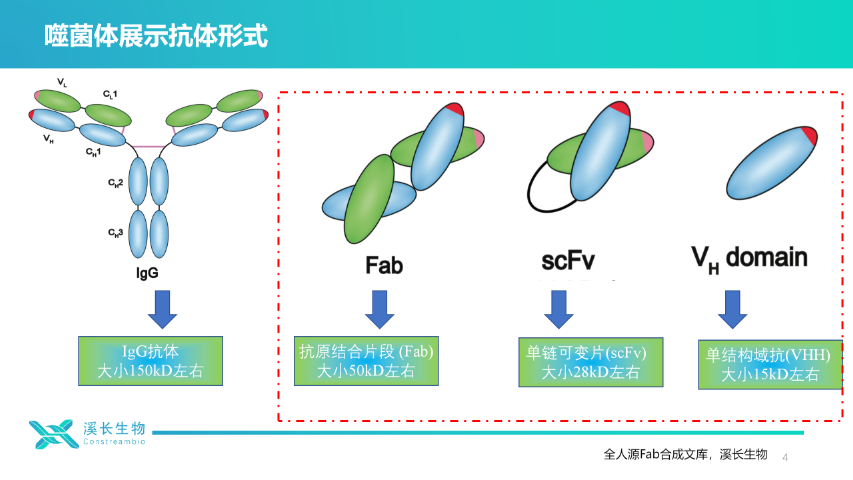
千亿级文库革新抗体发现全人源Fab合成文库打破免疫原性困局:传统鼠源抗体临床免疫原性高达5%-89% ,溪长生物依托Trimer引物随机化策略构建全人源Fab合成文库,CDR3长度与氨基酸分布模拟天然多样性 。库容已达1011(2026年将突破10¹²),经数十靶点验证亲和力达pM级,避免人源化抗体的框架区残留免疫风险 。适用于CAR-T、双抗等复杂结构开发,降低临床脱靶率。同时溪长生物也在不断地通过跨学科技术整合,正逐步攻克新的难点,推动全合成文库从“高通量筛选工具”向“高质量抗体发现平台”升级,为罕见病靶点、多特异性抗体等前沿领域提供关键技术支撑。上海溪长生物全人源Fab合成噬菌体文库,一个月即可筛选高亲和力分子!重庆合成噬菌体文库技术科普
全人源Fab合成噬菌体文库,上海溪长技术,满足科研不同需求。青海合成噬菌体文库流程解析
溪长生物的全人源Fab合成文库构建是针对CDR3区做了独特设计,作为抗体与抗原结合的关键区域,CDR3的长度与氨基酸组成直接决定亲和力上限。上海溪长生物技术有限公司采用三核苷酸随机化技术(TrimerPrimers),在文库构建时实现CDR3区6-25个氨基酸的全覆盖,并通过密码子简并性优化,使20种天然氨基酸的分布频率与人体天然免疫库高度一致(如酪氨酸占比9.2%、半胱氨酸占比1.8%)。这种“仿生学”设计确保文库中存在大量天然免疫系统难以产生的高亲和力构象。青海合成噬菌体文库流程解析